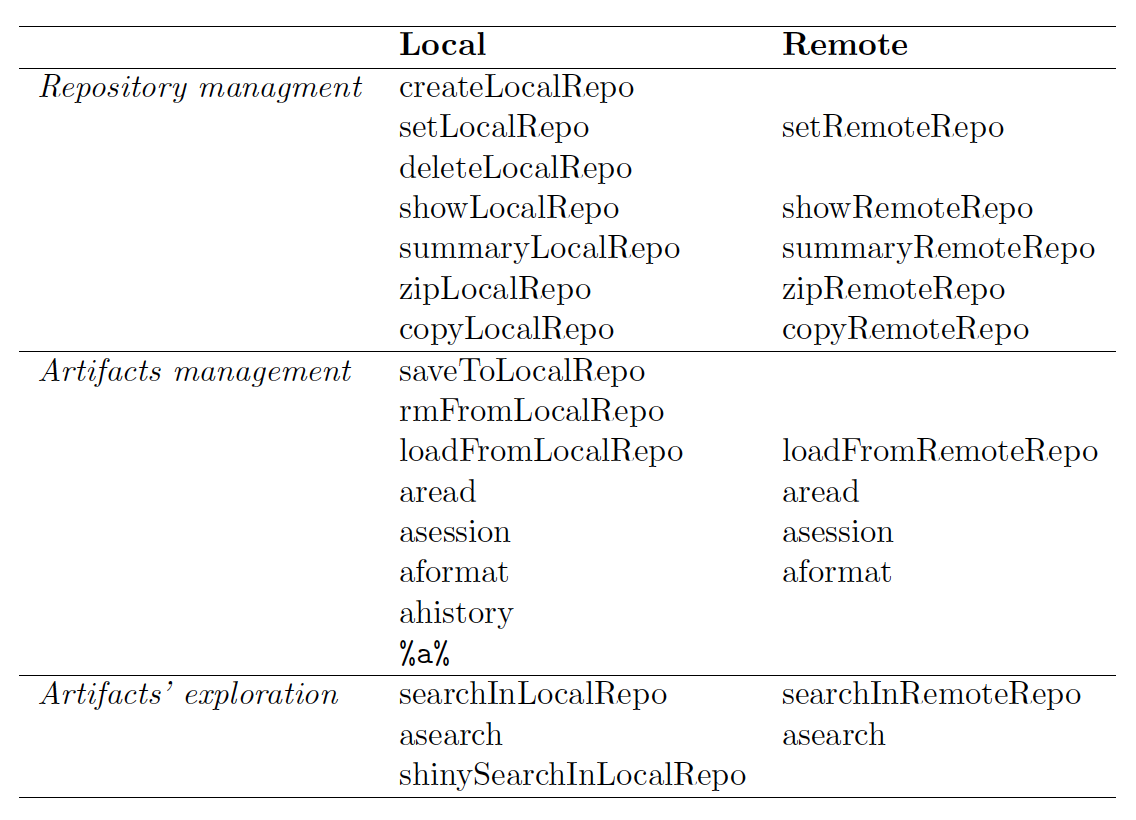
Jak serializować obiekty z użyciem pakietu archivist?
W programie R wszystko jest obiektem. Każdy model, tabela, wykres, funkcja, wszystko jest obiektem, który żyje w pamięci aktualnie uruchomionej sesji R.
Często jednak pojawia się potrzeba zapisania, odszukania lub przesłania obiektu z jednej sesji R do innej. Takie operacje są wspierane przez bibliotekę archivist.
Omówimy ją poniżej w oparciu o typowe przypadki użycia.
Kluczowe pojęcia
Funkcja skrótu MD5 - kryptograficzna funkcja przypisująca każdemu dowolnemu ciągowi bajtów jednoznacznie wyznaczony hash, będący 32 znakami cyframi szesnastkowymi (256 bitów). Więcej informacji: https://pl.wikipedia.org/wiki/MD5
GitHub/Bitbucket - archiwa kodu w modelu cloud oparte o system Git/Mercurial
Repozytorium - zbiór obiektów wraz z API zapewniającym dodatkowo operacje wyszukiwania, odczytywania i zapisywania obiektów.
Repozytoria dzielimy na lokalne (umieszczone na dysku) i zdalne (w pakietach R lub na GitHub/Bitbucket).
Tag - etykieta opisująca zawartość obiektu, ułatwia odszukiwanie obiektów o określonych cechach.
Odzyskiwanie obiektów
Obiekty, które są już w repozytorium można odczytać funkcją aread().
Przykładowo poniższa linia odczyta z repozytorium graphGallery użytkownika pbiecek na Github obiekt o hashu f05f0ed0662fe01850ec1b928830ef32. Jest to wykres wykonany w pakiecie ggplot2.
archivist::aread("pbiecek/graphGallery/f05f0ed0662fe01850ec1b928830ef32")
Aby odczytywać obiekty z lokalnego repozytorium, należy funkcją setLocalRepo() ustawić ścieżkę do repozytorium na dysku.
Następnie wystarczy wywołać funkcję aread() podając jako argument jedynie hash obiektu.
Poniżej odczytamy w ten sposób model liniowy.
library(archivist)
(repoDir <- system.file("graphGallery", package = "archivist"))
## [1] "/Library/Frameworks/R.framework/Versions/3.3/Resources/library/archivist/graphGallery"
setLocalRepo(repoDir)
aread("2a6e492cb6982f230e48cf46023e2e4f")
##
## Call:
## lm(formula = Petal.Length ~ Sepal.Length + Species, data = iris)
##
## Coefficients:
## (Intercept) Sepal.Length Speciesversicolor
## -1.7023 0.6321 2.2101
## Speciesvirginica
## 3.0900
Wyszukiwanie obiektów
Obiekty są zapisywane do bazy danych razem z tagami, które umożliwiają łatwe wyszukiwanie tych obiektów.
Przykładowe tagi to data utworzenia, klasa, nazwy zmiennych, informacje o zależnościach pomiędzy obiektami czy nazwa symbolu do którego przypisany jest obiekt.
Przykładowo, poniższa instrukcja odczytuje z repozytorium wszystkie obiektu klasy lm, które wśród predyktorów mają zmienną Sepal.Length.
models <- asearch("pbiecek/graphGallery",
patterns = c("class:lm", "coefname:Sepal.Length"))
lapply(models, coef)
## $`18a98048f0584469483afb65294ce3ed`
## (Intercept) Sepal.Length
## -7.101443 1.858433
##
## $`2a6e492cb6982f230e48cf46023e2e4f`
## (Intercept) Sepal.Length Speciesversicolor Speciesvirginica
## -1.7023422 0.6321099 2.2101378 3.0900021
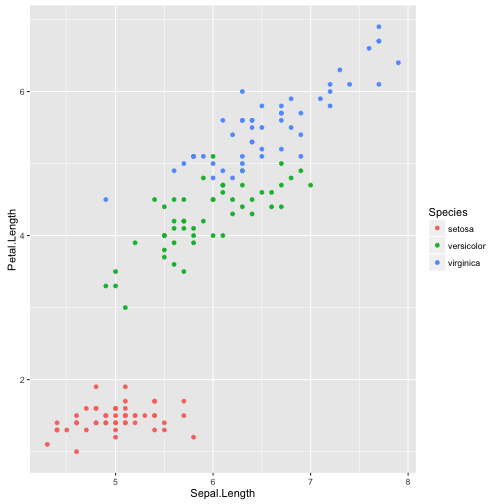
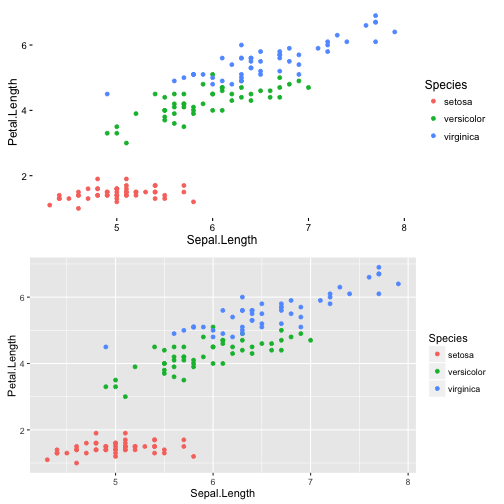
W podobny sposób można z lokalnego repozytorium odczytać wszystkie wykresy klasy gg, które na osi OX mają zmienną Sepal.Length,
Odczytajmy je i wyświetlmy na ekranie.
plots <- asearch(patterns = c("class:gg", "labelx:Sepal.Length"))
length(plots)
## [1] 2
library("gridExtra")
do.call(grid.arrange, plots)
Historia obiektu
W odtwarzalnych obliczeniach kuszącą obietnicą jest możliwość odtworzenia historii obiektu, szczegółowych informacji jak obiekt był zbudowany. Gdyby to było możliwe, dla każdego wyniku moglibyśmy sprawdzić jak został on osiągnięty.
Z pakietem archivist to często jest możliwe. Trzeba tylko używać operatora %a% przy tworzeniu obiektów. Ten operator zapisuje do repozytorium wszelkie informacje potrzebne do odtworzenia obiektu.
library("archivist")
library("dplyr")
createLocalRepo("arepo", default = TRUE)
## Directory arepo did not exist. Forced to create a new directory.
iris %a%
filter(Sepal.Length < 6) %a%
lm(Petal.Length~Species, data=.) %a%
summary() -> tmp
## Tracing lm(Petal.Length ~ Species, data = `__LHS`) on exit
Funkcją ahistory() można odtworzyć historię danego obiektu.
ahistory(tmp)
## env[[nm]] [ff575c261c949d073b2895b05d1097c3]
## -> filter(Sepal.Length < 6) [d3696e13d15223c7d0bbccb33cc20a11]
## -> lm(Petal.Length ~ Species, data = .) [990861c7c27812ee959f10e5f76fe2c3]
## -> summary() [050e41ec3bc40b3004bc6bdd356acae7]
ahistory(md5hash = "050e41ec3bc40b3004bc6bdd356acae7")
## env[[nm]] [ff575c261c949d073b2895b05d1097c3]
## -> filter(Sepal.Length < 6) [d3696e13d15223c7d0bbccb33cc20a11]
## -> lm(Petal.Length ~ Species, data = .) [990861c7c27812ee959f10e5f76fe2c3]
## -> summary() [050e41ec3bc40b3004bc6bdd356acae7]
Gdy obiekt jest zapisywany do repozytorium zapamiętywana jest też informacja o zainstalowanych pakietach. Można ją odtworzyć i odczytać.
sinfo <- asession("050e41ec3bc40b3004bc6bdd356acae7")
head(sinfo$packages)
## package * version date source
## archivist * 2.0.4 2016-05-13 CRAN (R 3.3.0)
## assertthat 0.1 2013-12-06 CRAN (R 3.3.0)
## BetaBit * 1.2.1 2016-05-26 CRAN (R 3.3.0)
## bitops 1.0-6 2013-08-17 CRAN (R 3.3.0)
## chron 2.3-47 2015-06-24 CRAN (R 3.3.0)
## colorspace 1.2-6 2015-03-11 CRAN (R 3.1.3)
Tworzymy repozytorium i wypełniamy je obiektami
Aby zbudować lokalne repozytorium można wykorzystać funkcję createLocalRepo().
Utwórzmy repozytorium arepo w lokalnym katalogu i uczyńmy je od razu domyślnym.
repo <- "arepo"
createLocalRepo(repoDir = repo, default = TRUE)
Zbudujmy przykładowy wykres i dodajmy go do repozytorium używając funkcji saveToLocalRepo(). Repozytorium domyślne jest określone, nie ma więc konieczności podawania go w funkcji saveToRepo().
library("ggplot2")
pl <- qplot(Sepal.Length, Petal.Length, data = iris)
saveToLocalRepo(pl)
## [1] "abf399fa855697165294a45a6a2280f3"
## attr(,"data")
## [1] "ff575c261c949d073b2895b05d1097c3"
Hash obiektu jest produkowany funkcją digest(). Możemy zawsze sprawdzić czy się zgadza dla odczytanego obiektu.
digest::digest(pl)
## [1] "abf399fa855697165294a45a6a2280f3"
Funkcjami showLocalRepo() i summaryLocalRepo() można wyświetlić statystyki dotyczące obiektów w repozytoriach.
showLocalRepo(method = "tags")
## artifact
## 1 abf399fa855697165294a45a6a2280f3
## 2 abf399fa855697165294a45a6a2280f3
## 3 abf399fa855697165294a45a6a2280f3
## 4 abf399fa855697165294a45a6a2280f3
## 5 abf399fa855697165294a45a6a2280f3
## 6 abf399fa855697165294a45a6a2280f3
## 7 abf399fa855697165294a45a6a2280f3
## 8 a2bdc39c3b30379a9b009970ec06d606
## 9 abf399fa855697165294a45a6a2280f3
## 10 ff575c261c949d073b2895b05d1097c3
## 11 ff575c261c949d073b2895b05d1097c3
## 12 ff575c261c949d073b2895b05d1097c3
## 13 abf399fa855697165294a45a6a2280f3
## tag createdDate
## 1 format:rda 2016-05-29 23:57:47
## 2 name:pl 2016-05-29 23:57:47
## 3 class:gg 2016-05-29 23:57:47
## 4 class:ggplot 2016-05-29 23:57:47
## 5 labelx:Sepal.Length 2016-05-29 23:57:47
## 6 labely:Petal.Length 2016-05-29 23:57:47
## 7 date:2016-05-29 23:57:47 2016-05-29 23:57:47
## 8 format:rda 2016-05-29 23:57:47
## 9 session_info:a2bdc39c3b30379a9b009970ec06d606 2016-05-29 23:57:47
## 10 format:rda 2016-05-29 23:57:47
## 11 format:txt 2016-05-29 23:57:47
## 12 relationWith:abf399fa855697165294a45a6a2280f3 2016-05-29 23:57:47
## 13 format:png 2016-05-29 23:57:47
summaryLocalRepo()
## Number of archived artifacts in Repository: 1
## Number of archived datasets in Repository: 1
## Saves per day in Repository:
## Saves
## 2016-05-29 3
Inne zastosowania
W połączeniu z funkcją trace() możemy dodać archiwizację wszystkich obiektów tworzonych przez określoną funkcje. Np. możemy do archiwum zapisać wszystkie modele liniowe zbudowane funkcją lm().
library("archivist")
createLocalRepo("allModels", default = TRUE)
## Directory allModels did not exist. Forced to create a new directory.
trace(lm, exit = quote(saveToLocalRepo(z)))
## Tracing function "lm" in package "stats"
## [1] "lm"
Lub możemy połączyć pakiet archivist z pakietem knitr i wszystkie obiekty określonej klasy wyprodukowane przez knitra dodać do repozytorium.
Funkcja addHooksToPrint () automatycznie dodaje uchwyty w podpisach rysunków, jest to bardzo wygodne do odzyskiwania obiektów.
Przykład działania tego połączenia można obejrzeć pod adresem http://bit.ly/1nW9Cvz
addHooksToPrint(class=c("ggplot", "data.frame"),
repoDir = "arepo", repo = "Eseje", user = "pbiecek", subdir = "arepo")
Dla repozytoriów można też przygotować listę wszystkich obiektów tam umieszczonych wraz z ich tagami. Przykład takiego katalogu znajduje się na stronie https://github.com/pbiecek/Eseje/tree/master/arepo